Künstliche Intelligenz beschleunigt MRI des Blutflusses
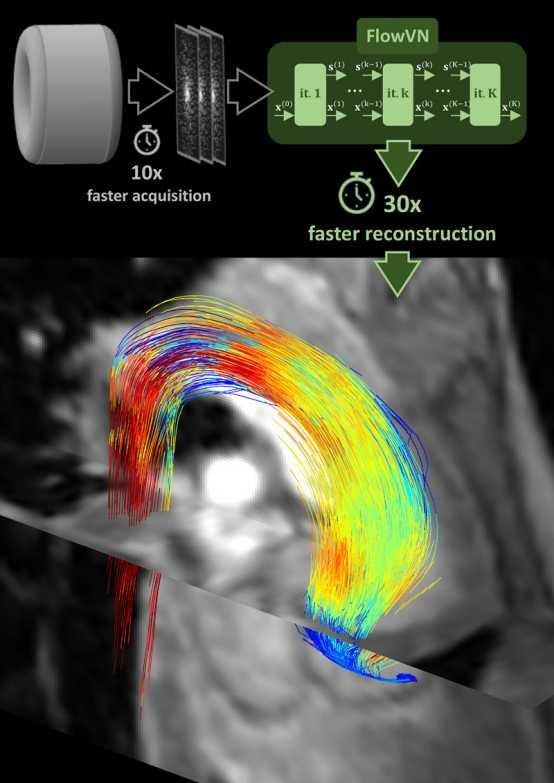
«Dank dieses Verfahrens könnte die quantitative Magnetresonanztomografie enorme Fortschritte machen», freut sich Sebastian Kozerke, Professor für Biomedizinische Bildgebung an ETH und Universität Zürich. Zusammen mit Valery Vishnevskiy und Jonas Walheim hat er eine Methode entwickelt, die das sogenannte 4D-Fluss-MRI massiv beschleunigt.
«Heute dauern Aufnahme und Nachverarbeitung eines 4D-Fluss-MRIs bis zu 30 Minuten. Unsere Resultate zeigen, dass dies in Zukunft innerhalb von fünf Minuten möglich sein könnte.» Die zu Grunde liegende Arbeit ist Anfang dieser Woche im Fachjournal «Nature Machine Intelligence» als Artikel und Cover der April-Ausgabe erschienen.
Die Magnetresonanztomografie (MRT oder MRI) ist ein wichtiges Verfahren für die klinische Diagnose. Sie ist nicht gesundheitsschädlich und liefert präzise Bilder aus dem Körperinneren. Mit der Methode können Weichteile wie Gewebe und Organe in 3D und mit hohem Kontrast dargestellt werden.
Darüber hinaus liefern spezielle Aufnahmetechniken Informationen über die Dynamik des Herzkreislaufsystems. Insbesondere erlauben 4D-Fluss-MRI Messungen, dynamische Veränderungen des Blutflusses in den Blutgefässen zu quantifizieren. Solche dynamischen Bilder sind insbesondere zur Erkennung von Herz-Kreislauf-Erkrankungen von grossem Nutzen.
Das heute gängige 4D-Fluss-MRI hat jedoch einen gewichtigen Nachteil: die Methode ist sehr zeitaufwändig. Zwar kann die Datenaufnahme im MRI-Scanner bereits innerhalb von vier Minuten erfolgen. Jedoch hat diese komprimierte Erfassung (sogenanntes Compressed Sensing) ihren Preis: Die folgende Bildrekonstruktion erfolgt iterativ und dauert darum sehr lange.
Bis die Bilder auf dem Rechner des Arztes sind, vergehen 25 Minuten und mehr. Entsprechend liegen die Ergebnisse der Messung erst lange, nachdem der Arzt die Untersuchung abgeschlossen hat, vor. Aus diesen Gründen hat sich das 4D-Fluss-MRI im medizinischen Alltag noch nicht durchgesetzt. Veränderungen von Blutflussströmungen werden heute primär mittels Ultraschall diagnostiziert – eine schnellere, aber im Vergleich zum MRI weniger präzise Methode.
Elegante und effiziente Algorithmen
Im eben veröffentlichten Fachartikel zeigen die Forscher von ETH und Universität Zürich einen Weg auf, wie die Bildrekonstruktion für das 4D-Fluss-MRI schneller und damit praxistauglicher gemacht werden kann. „Die Lösung sind elegante und effiziente Algorithmen auf der Basis neuronaler Netzwerke“, erklärt Kozerke.
Vishnevskiy, Kozerke und Walheim nennen ihren neuen Ansatz FlowVN. Dieser basiert auf maschinellem Lernen, konkret auf sogenanntem Deep Learning. Die Software lernt aus Daten, mit denen sie vorgängig trainiert wird. Das Besondere an FlowVN ist die Effizienz, denn die Methode kombiniert Training mit dem Vorwissen über die Aufnahmetechnik.
Das heisst, dass nicht Tausende von Trainingsbeispielen benötigt werden, sondern auf der Basis von wenigen Daten generalisiert wird. «Das Netzwerk braucht also nur sehr wenig Training, um verlässliche Ergebnisse zu liefern», erklärt Vishnevskiy.
Dass das funktioniert, konnten die Forscher in der eben erschienenen Arbeit zeigen. Sie trainierten die Software mit 11 MRI-Scans von gesunden Probanden. Diese Daten genügten, um innerhalb von nur 21 Sekunden auf einem gewöhnlichen Rechner pathologischen Blutfluss in der Aorta eines Patienten präzise wiederzugeben. Das Verfahren war damit um ein Vielfaches schneller als herkömmliche Methoden – und lieferte erst noch bessere Resultate.
Der klinischen Diagnostik einen Schub verleihen
«Wir hoffen, dass FlowVN dem Einsatz von 4D-Fluss-MRI in der klinischen Diagnostik einen Schub verleihen wird», sagt Kozerke. Für die vorliegende Studie wurden die Daten offline rekonstruiert. Der nächste Schritt für die Zürcher Forscher wird deshalb die Installation der Software auf klinischen MRI-Geräten sein.
«Danach fassen wir grössere klinische Patientenstudien ins Auge», so Kozerke. Zu Gute kommt den Wissenschaftlern dabei die langjährige Partnerschaft mit der Radiologie und Kardiologie am Universitätsspital Zürich.
Bestätigen die Folgeuntersuchungen die Resultate von Kozerkes Team, könnte die Methode dereinst Eingang in den klinischen Alltag finden. «Bis dahin wird es aber mindestens noch vier bis fünf Jahre dauern», schätzt Kozerke.
Um den wissenschaftlichen Prozess zu beschleunigen, hat sein Team die lauffähigen Codes mitsamt Datenbeispielen Open Source zur Verfügung gestellt. Auf diese Weise können andere Wissenschaftler die Methode testen und reproduzieren.
Literaturverweis
Vishnevskiy V, Walheim J, Kozerke S. Deep variational network for rapid 4D flow MRI reconstruction. Nature Machine Intelligence, 13. April 2020. doi: 10.1038/s42256-020-0165-6
ETH Zürich
Rämistrasse 101
CH8092 Zürich
Telefon: +41 (44) 63211-11
Telefax: +41 (44) 63210-10
http://www.hk.ethz.ch
Professor für Biomedizinische Bildgebung an ETH und Universität Zürich
Telefon: 41 (76) 340 5470
E-Mail: kozerke@biomed.ee.ethz.ch
![]()





